普通转录组(Bulk RNA)是指生物组织样品在一定时间的全部 mRNA的转录情况,获得的是一个大的细胞群体中单个基因的平均表达水平,可以用来比较不同组织间的表达差异,但无法展示个细胞的转录表达水平;单细胞转录组测序(Single cell RNA sequencing,scRNA-seq)在单个细胞水平上构建每个细胞的表达谱,能够揭示单个细胞的基因表达状态,反映细胞间的异质性,但在将实体组织解离成单个细胞的过程中,丢失了空间信息,而空间异质性是器官功能的关键特征;空间转录组学(Spatially Resolved Transcriptomics,SRT)的出现,能够把细胞在组织分布的位置信息和基因表达信息相结合,在空间背景下了解细胞异质性。
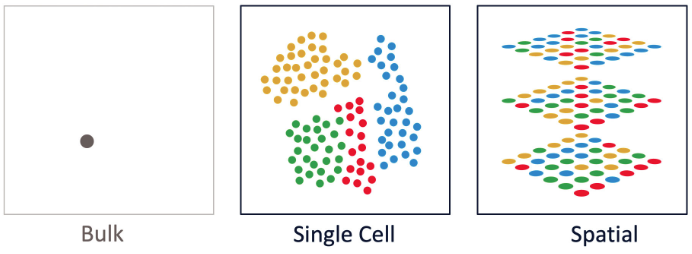
图1. bulk转录组、单细胞转录组和空间转录组差别
空间转录组的概念最早来源于FISH(fluorescently-labeled RNA sequences),FISH是带有荧光的探针与目标结合实现靶基因的原位检测,从而实现定性定量和相对位置的分析,目前FISH仍然是临床诊断的常用技术之一。空间转录组技术经过最近几年的发展,在分辨率和通量上达到了大幅的提升,并于2020年被Nature Methods评为年度技术方法,发文量也急剧上升。
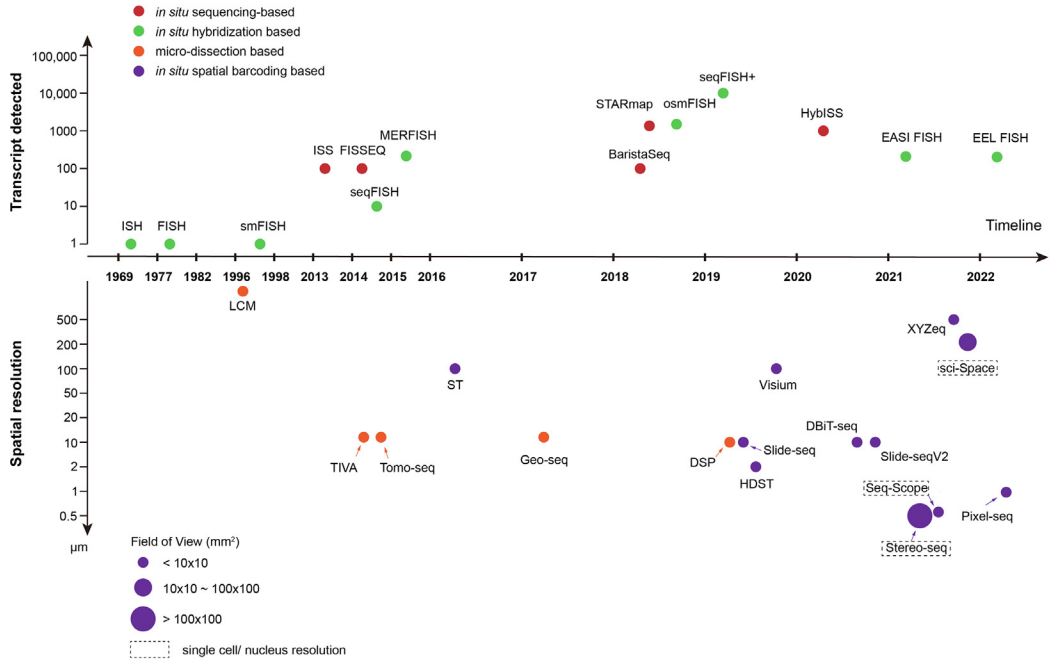
图2. 空间转录组学技术发展
根据原理的不同,SRT技术可分为基于成像和测序两大类。首先基于成像的SRT技术主要指显色基团与探针结合后通过显微镜对mRNA进行原位成像,该技术能直接原位反映目的基因分布,根据mRNA种类的不同可分为原位杂交(in situ hybridization, ISH)或原位测序(in situ sequencing, ISS),主要方法有seqFISH(Sequential Fluorescence In Situ Hybridization)和MERFISH(Multiplexed error-robust fluorescence In situ hybridization)。基于测序的SRT技术,不需要先验的基因序列知识,从组织中提取mRNA同时保留空间信息,随后通过高通量测序技术(next generation sequencing, NGS)对mRNA进行检测和分析;根据获取空间位置的方式不同,可分为基于直接捕获的显微切割法(如GEO-seq, LCM-Seq),和基于原位条形码法(如ST,Stereo-seq等),基于测序的SRT技术分辨率和捕获效率发展非常快。
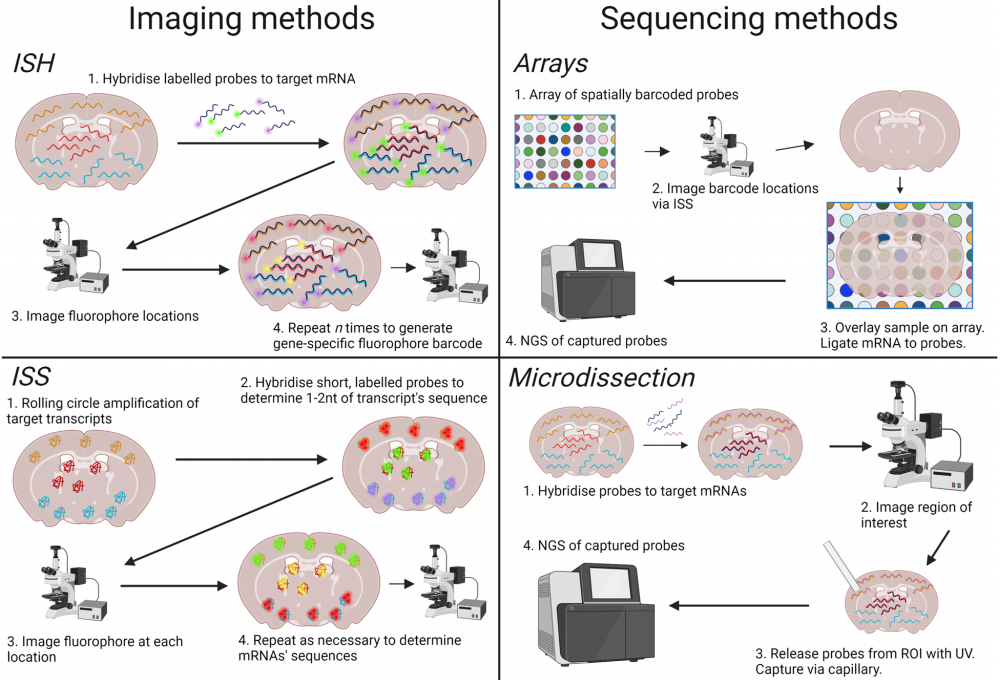
图3. 空间转录组学技术分类
尽管当前的空间转录组学技术的mRNA捕获效率和空间分辨率有待进一步提高,但鉴于其能解决空间结构问题,已被广泛应用于胚胎发育的时空组学图谱构建、分子和细胞分辨下重建3D器官、疾病机制和植物的细胞景观等方面的研究。
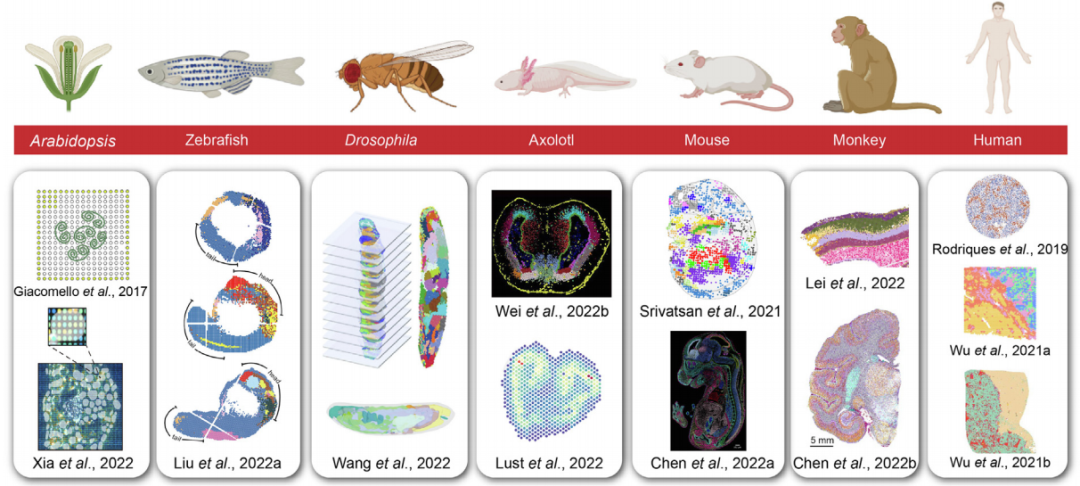
图4. 空间转录组技术应用
空间转录组能用于任何含有活性mRNA的样本,具体到每个项目需要考虑以下几个要素:
1)物种及组织类型,是否在可研究的范围内:不同的组织具有不同的属性,在某些情况下需要优化相关技术参数;
2)组织中RNA质量可能影响空间转录组的选择;
3)根据研究目的及各技术的优劣势选择具有合适检测范围及分辨率的方法,如ISH和ISS分辨率和mRNA捕获效率高,适用于假设检验实验,而包括Visium和STomics在内的芯片方法适用于无偏假设生成和图谱生成的研究;

图5. 空间转录组设计指南
参考文献:
