多组学关联分析是一种综合多种不同组学数据来了解生物体的方法,包括基因组、转录组、蛋白质组、代谢组和表型组等等。通过整合不同水平的结果,关联分析有助于系统性地研究疾病发生发展机理、寻找生物标志物和进行疾病的早期诊断,从而更好地进行个体化治疗。
当拥有多种组学数据时,选择哪些分析方法将不同来源的数据关联在一起,并以简洁、直观的图形表示出来可是一大难题。
今天就来学习一下在高分文章中如何关联多组学的数据吧~
PCA分析是组学研究者再熟悉不过的统计方法,在高分文章中也频频出现它的身影。
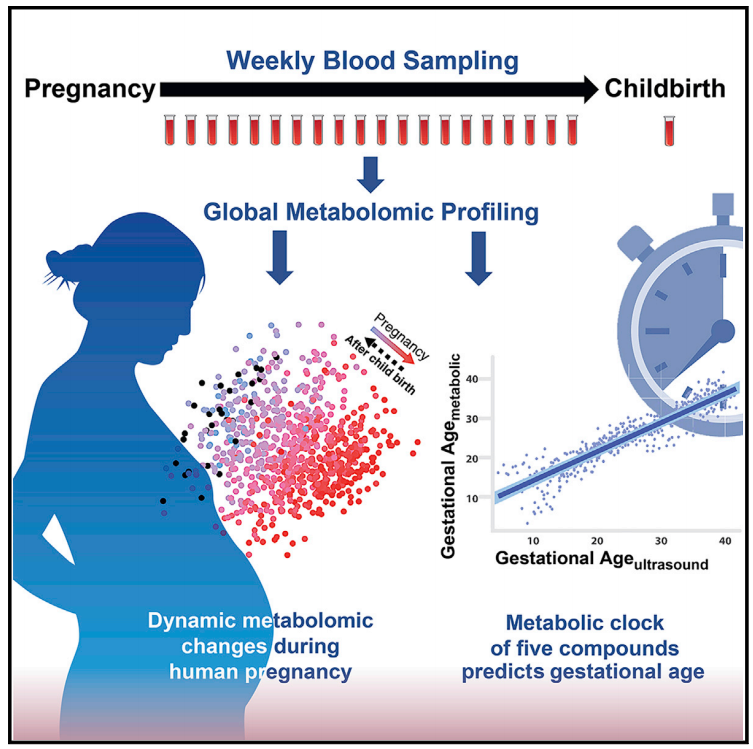
图1. 妊娠时间与血液代谢组学PCA图
2020年发表于Cell的"Metabolic Dynamics and Prediction of Gestational Age and Time to Delivery in Pregnant Women"一文中,作者通过对孕期女性在不同怀孕时期的血液样本进行代谢组学检测,分析了孕妇在整个孕期的生理特点与代谢变化。
文章开篇就是作者针对血液代谢组学绘制的主成分分析图,整合了代谢组学与临床表型信息。图中样本随着妊娠时间不同呈现出从蓝到红的颜色过渡,可见样本随妊娠时间呈现出了一定分布变化趋势,说明在不同妊娠时间中,孕妇血液代谢谱发生了显著而有规律的变化,以此作为引入便可在后续开展围绕不同妊娠时间具体代谢物变化的分析。
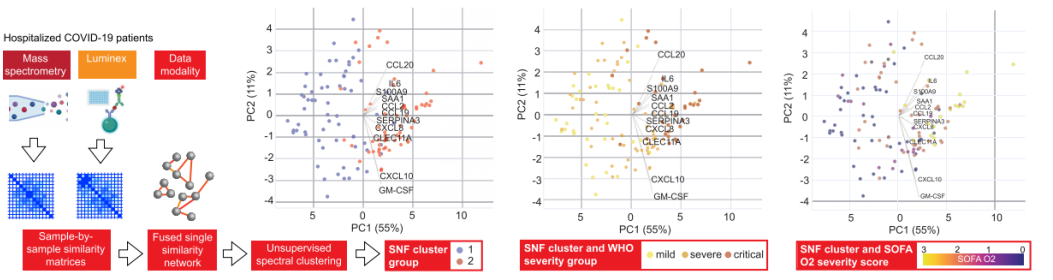
图2. 血浆蛋白组根据分型、疾病严重程度和SOFA氧评分分析的PCA图
2022年Cell上的"A blood atlas of COVID-19 defines hallmarks of disease severity and specificity"一文,作者同样通过复数PCA图呈现样本多角度信息。研究中构建了COVID-19血浆蛋白样本之间相似度矩阵,推导出两种疾病分型。通过比较分型与疾病严重程度的无监督PCA结果,表明该分型可基于WHO分类标准的重症病例分为轻型病例较多组和危重型病例聚集组。此外发现两个与疾病严重程度的临床衡量指标,激发氧浓度和SOFA氧评分,PCA图中可见疾病越严重,SOFA氧评分越高,样本点颜色越偏黄色。
主成分分析(principal component analysis, PCA)是一种应用广泛的降维算法,能够去除噪声和不重要的特征,有效地利用少量的主成分使得数据的维度降低,从而有效地突显代谢组学数据的总体分布趋势以及组间样本的差异程度。图中样本点的距离越近,代表样本中代谢物的表达模式越相近。样本的距离越远,代表样本之间的差异越大。
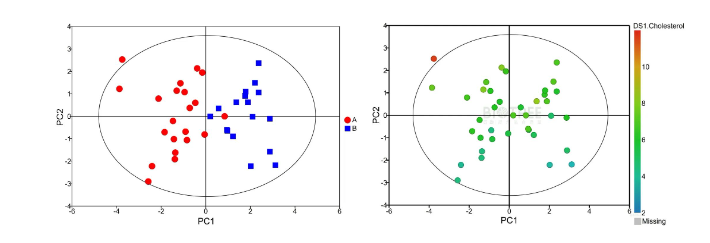
图3. 高血压代谢组学PCA图
样本图例的颜色除了可以反映组别聚类以外,在多组学的应用中,也可以用于反映具体一个组学变量,或者实验相关因素,如临床因素、环境因素等等。将变量的表达量作为样本点的颜色深浅反映在图中,展现出样本组中该变量表达的变化趋势,在一定程度上更加直观地反映出所选变量与样本的关联。
微生物组和代谢组学做关联分析,如何在揭示菌群样本的分布特征同时,也挖掘菌群、样本和代谢物三者之间或者两两之间的相关关系?
典型相关分析(Canonical Correlation Analysis, CCA),它能够同时反映菌群的分布特点与分析变量之间的内在联系。其基本思想和主成分分析非常相似。
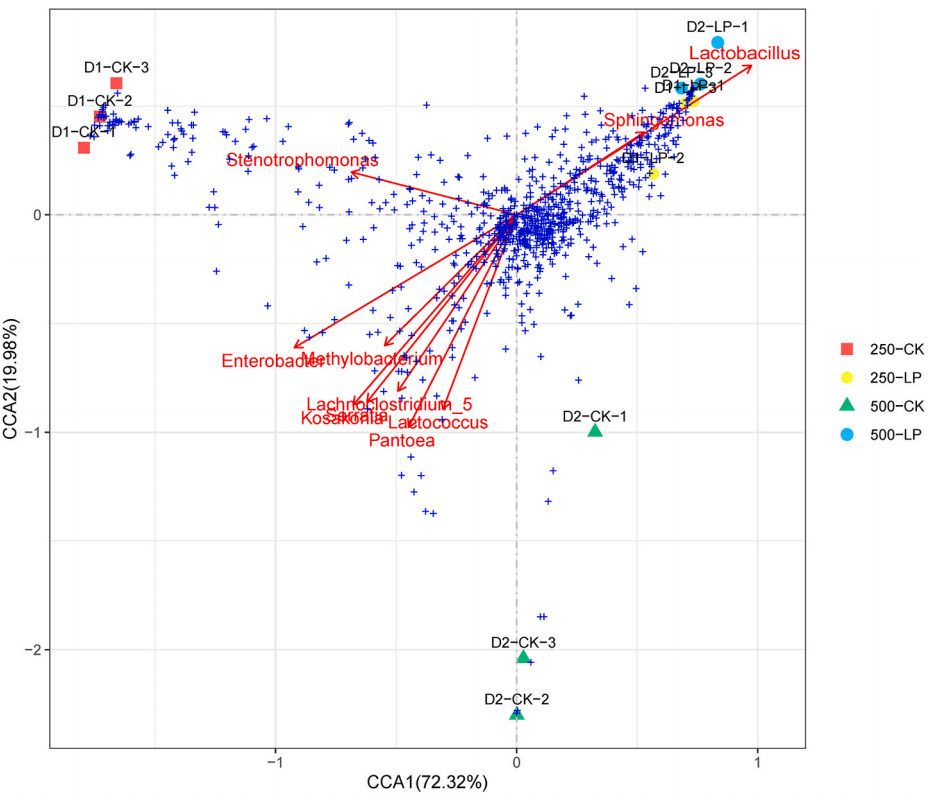
图4. 青贮构树饲料中菌群和代谢物的CCA分析
2021年在Journal of Cleaner Production上发表的"Chemical and bacterial composition of Broussonetia papyrifera leaves ensiled at two ensiling densities with or without Lactobacillus plantarum"文章中,作者通过CCA分析研究青贮饲料中细菌群落与代谢物的关系。图中红色箭头代表不同的菌群,箭头越长说明该菌群对代谢物的影响程度越大。蓝色点代表不同代谢物,不同颜色的几何形状代表样本。夹角大小代表菌群与代谢物间的正、负相关关系,夹角为锐角时,表示微生物与代谢物之间呈正相关,钝角时呈负相关,直角代表两者无相关性。结果显示,代谢物与细菌群落表现出相关趋势,特别是鞘氨醇单胞菌和乳酸菌在影响代谢物成分中起到关键作用。
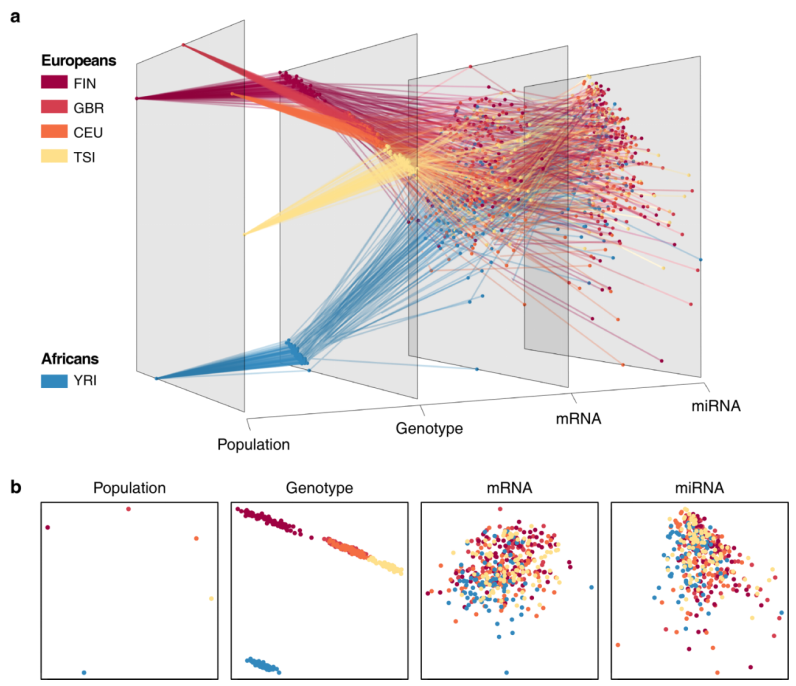
图5. 4种不同多组学数据的Grimon可视化
像上面所提到的常见二维图,如PCA、t-SNE等降维算法,只能突出单个水平的样本分布数据,而没有关于多个水平数据中复杂相互作用的线索。如何呈现多水平数据之间的分布联系?
"Grimon: graphical interface to visualize multi-omics networks"文章提出Grimon分析(Graphical interface to visualize multi-omics networks, Grimon),能够将多个二维平行坐标扩展到三维空间,有效地可视化多水平高维数据集。进行多组学分析时,Grimon分析可以展示每个样本在不同组学上的聚散程度,帮助我们更加直观地探索数据,从而理解多组学之间的复杂连接关系。
上图中使用淋巴母细胞样细胞系包括人群分组、基因组、RNA组和MicroRNA组四个水平数据,进行了多组学数据的Grimon可视化。连线颜色对应样本来源的人群,横轴代表多层水平。通过连线连接从群体到基因型和转录组不同水平的相同样本,可以从不同水平上,看到样本的聚散情况和位置变化。
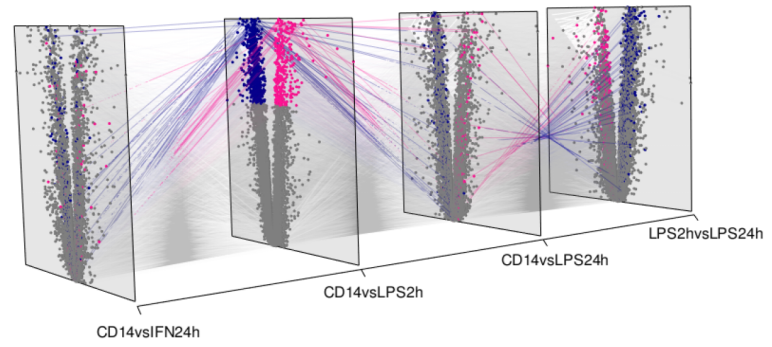
图6. 4种火山图数据的Grimon可视化
更进一步,Grimon的应用可不局限于可视化PCA或t-SNE结果。在解释多个对比策略的数据集时,图6中每个平面对应一个火山图,比较不同对比策略之间的联系。每条连线在不同的对比策略中连接同一变量,如代谢物、基因、蛋白等,Grimon可以有效地强调其中一种对比策略中的差异变量在其他对比策略中是如何相对分布的。
热图(Heatmap)通过将数据矩阵中的数值按照一定规律以颜色进行展示,利用颜色变化来可视化数据的高低。这种方法可以很直观地呈现多样本多变量的整体表达量变化,同时还可以展现聚类关系。不过多组学研究中所包含的数据信息往往很多,单组学热图所能够呈现的内容已经无法满足我们的需要。
Mulheatmaps多维数据热力图是非常合适的解决方法。它能够在同一张图片上呈现多个组学的热图和注释,关联多种类型因素。而且多个组学数据用同一个含量卡尺进行度量,更适用于样本比较。
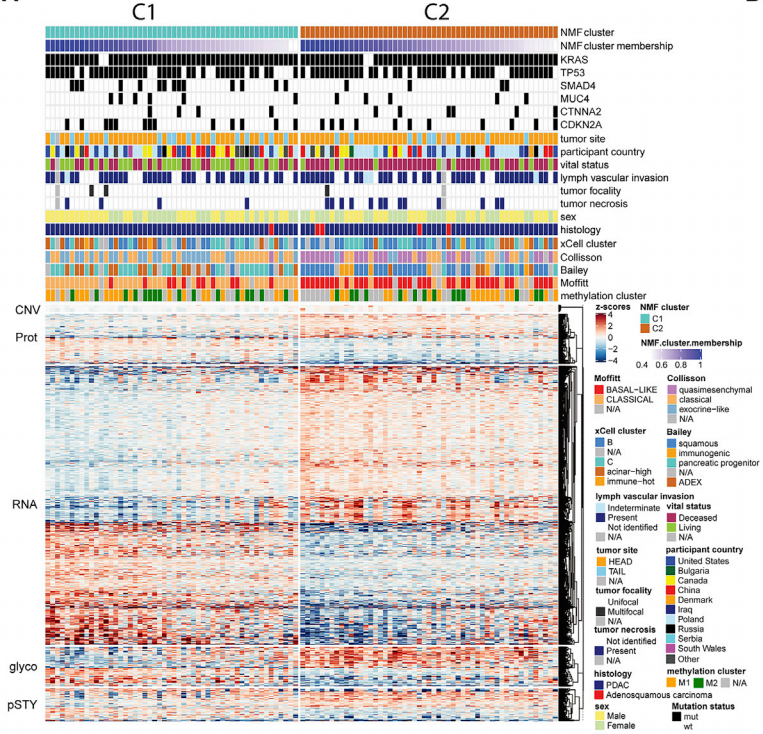
图7. 胰腺导管腺癌样本多维数据热力图
2021年发表于Cell的"Proteogenomic characterization of pancreatic ductal adenocarcinoma",热图呈现胰腺导管腺癌样本的蛋白质组、RNA组、糖蛋白组、临床因素和样本分型等多水平信息。主体部分横坐标为样本,纵坐标为组学变量,色块呈现对应变量的表达水平,以同一标准的红蓝色表示高低,颜色越红,说明表达量越高,颜色越蓝,说明表达量越低。色块一侧的树状图展现聚类结果。热图侧面根据研究目的需要,排列展示了样本的其他信息与注释内容。
最后介绍加权基因共表达网络分析(Weighted correlation network analysis, WGCNA),这是一种分析多组样本表达模式的分析方法,将表达相似的因素进行聚类,可用于探索聚类模块网络与关键因素之间的联系。
2020年发表在PNAS上的"Multi-omics analysis on an agroecosystem reveals the significant role of organic nitrogen to increase agricultural crop yield"整合了代谢组学、离子组学、微生物组学及表型组学等多水平数据,揭示了农业生态系统对不同耕作措施的响应特征并确定了土壤日晒是促进作物产量的关键因素,并阐明有机氮在农业生产中的关键地位。
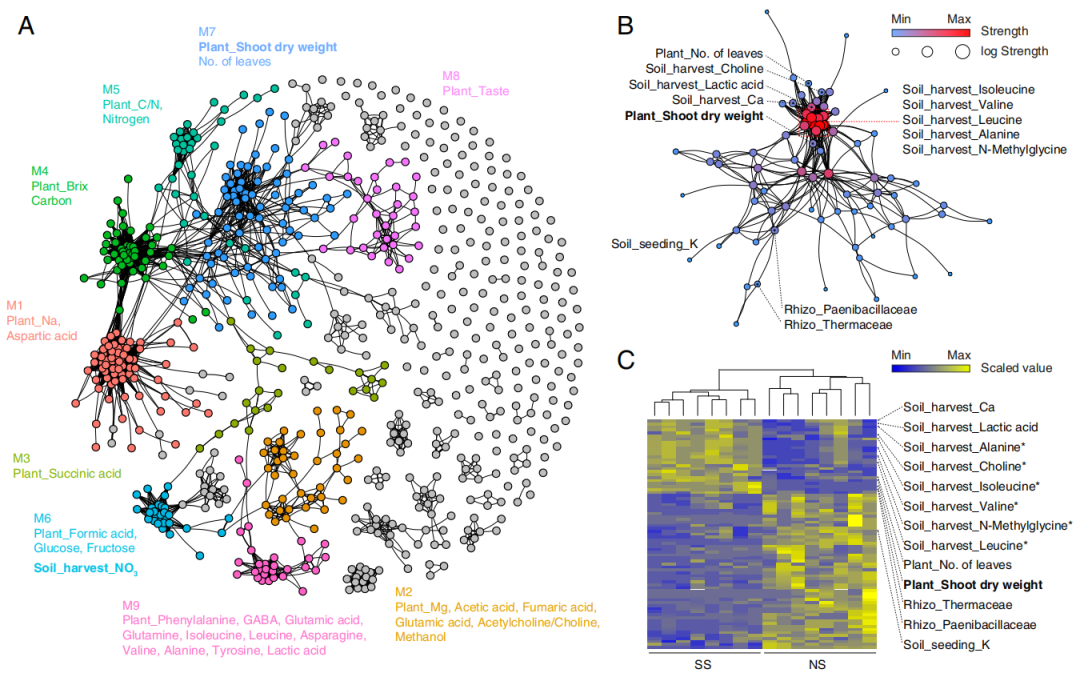
图8. 农业生态系统的WGCNA分析
该研究还通过WGCNA分析植物表型、植物代谢组、土壤代谢组、土壤离子组和根际微生物组等多水平数据构建相关网络,节点和连线分别表示组学变量和变量之间的相关性。农业生态系统中的复杂相互作用表现出植物性状与土壤代谢物、矿物质、微生物等相关的9个网络模块(从M1到M9),并用不同颜色表示。结果表明M7中的土壤有机氮是与植物产量高度相关的关键代谢产物。
一篇精彩的组学文章,必然是从多组学、多角度对研究内容进行描述,再配合使用关联分析方法和美观的可视化图达到更好的效果。
今天的分享就到这里,小编祝大家都能写出满意的文章~